高效利用现有数据及公共数据库发表SCI实例
对于广大科研工作者来说,有两个问题时常困扰心头。
第一,如何充分利用实验室现有的科研数据;
第二,如何有效利用公共数据库结果,结合生物信息学分析,充实自己的科研发现,最终发表一篇满意的SCI文章。
今天,小编根据自己刚发表在BMC Genomics的文章MiR-190a potentially ameliorates postoperative cognitive dysfunction by regulating *Tiam1*(IF = 3.501),实例讲解如何结合公共数据库数据,发表SCI文章,希望可以给大家带来帮助。
我们要研究的疾病是“术后认知障碍症”(POCD),该疾病是老年人手术后出现中枢神经系统并发症,表现为精神错乱、焦虑、人格的改变以及记忆受损。被疾病困扰的患者会出现人格、社交能力及认知能力和技巧的变化称为手术后认知功能障碍。
首先简单介绍一下实验中包含的四部分数据:
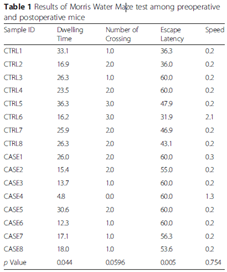
A. 健康组和POCD模型小鼠的生理实验指标(n = 16)以及水迷宫实验结果;
B. 健康组和POCD模型小鼠(n = 6)海马体microRNA芯片测序数据;
C. 公共数据库验证数据以及target miRNA分子机制验证数据;
D. qrt-PCR数据结果;
根据水迷宫实验结果(表1)表明,POCD模型小鼠在“停留时间”和“逃避潜伏期”和健康组相比存在显著的差异,证明POCD模型小鼠的记忆力显著的下降。
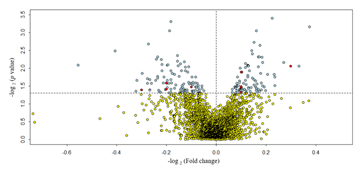
使用海马体microRNA芯片测序数据(芯片类型:Affymetrix miRNA 4.0),进行了差异分析(图1),在两组之间,我们找到了一些可能导致POCD症状发生的靶向microRNA。但是由于microRNA在脑区表达量比较低、样本数量比较少等原因,差异分析结果有可能存在假阳性的结果。为了增加分析结果的可靠性,我们使用了GEO数据库中的microRNA芯片测序数据,对分析结果进行了验证。在公共数据库中筛选验证数据时,应当选择类似的实验设计的数据和相同的测序平台,以保证实验结果的准确性。之后,我们使用qrt-PCR验证靶向microRNA的结果。
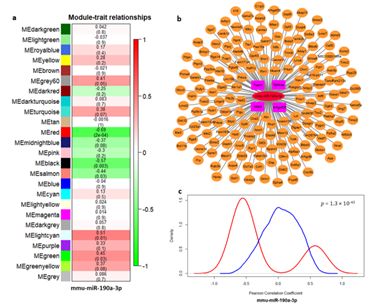
当找到了对表型有重要作用的microRNA以后,那么下一步一定是去探索该microRNA是通过什么样的生物学机制,调控了哪些重要的基因,从而达到对表型的影响。但是遗憾的是,我们手头并没有这些样本的转录组测序数据,没有办法进一步探索我们感兴的MiR-190a的生物学机制了。但是,好在有一个极其方便的工具,它叫做GEO数据库。在GEO数据库中,通过检索,找到了既有microRNA测序数据,又有mRNA测序数据的一批小鼠海马体测序样本。将该数据的原始数据下载下来,通过重新的mapping和alignment,我们得到了该数据的表达矩阵。通过共表达分析(图2),找到了和MiR-190a最具有相关性的一组基因集合,而这些基因在表达水平来看都有可能是MiR-190a潜在的作用靶向基因。通过相关性系数分布的分析可以发现,在整体基因组水平来看,基因和MiR-190a的相关性基本上是正相关、负相关各一半的。但是和MiR-190a显著相关的这个基因集合,显著呈现负相关(P = 1.3 E-10)。负相关的结果,也符合microRNA和靶基因的一般生物学规律。
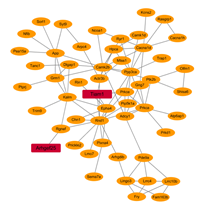
通过通路的富集分析,我们发现MiR-190a主要调控轴突引导、钙离子通道等一些在精神类疾病中起到重要作用的通路上。之后,结合蛋白质相互作用的数据库STRING(https://string-db.org),我们把MiR-190a的靶向基因范围进一步缩小到Tiam1、Arhgef25上(图3)。而Tiam1在之前的精神类疾病中,都反复的证明了该基因有重要的生物学作用。
通过系统的分析后,我们基本上阐述清楚MiR-190a的生物学功能和生物学机制。巧妙的利用了公共数据库的作用,充实我们自身的实验结果,从而夯实有限的数据结果,得以发表该文章。